
GIẢI TRÌNH TỰ METAGENOMICS
Metagenomics nghiên cứu tập hợp bộ gene của tất cả sinh vật trong mẫu môi trường. Metagenomics được ứng dụng nhiều trong nghiên cứu hệ vi sinh vật trong môi trường mà không cần phân lập, nuôi cấy những loài riêng lẻ. Công nghệ này có thể được ứng dụng trong nhiều mẫu môi trường đa dạng như đất, nước, biển, bùn, rễ cây, ruột, phân hay cơ thể động vật,...
Metagenomics trả lời 2 câu hỏi: vi sinh vật nào đang ở đó, và chúng đang làm gì?
Tiềm năng của vi sinh vật có thể giải quyết được các vấn đề thế giới đang đối mặt là rất lớn và chưa được khai phá hết. Các vấn đề từ kháng kháng sinh, bệnh nhiễm, năng suất trồng trọt, chăn nuôi, ô nhiễm môi trường và biến đổi khí hậu đều có liên quan đến biến đổi quần xã vi sinh vật. Chính vì vậy, hiểu về thế giới của vi sinh vật, chúng ta có thể mở ra tiềm năng của chúng để cải thiện cuộc sống của chính chúng ta!
CÁC DẠNG METAGENOMICS
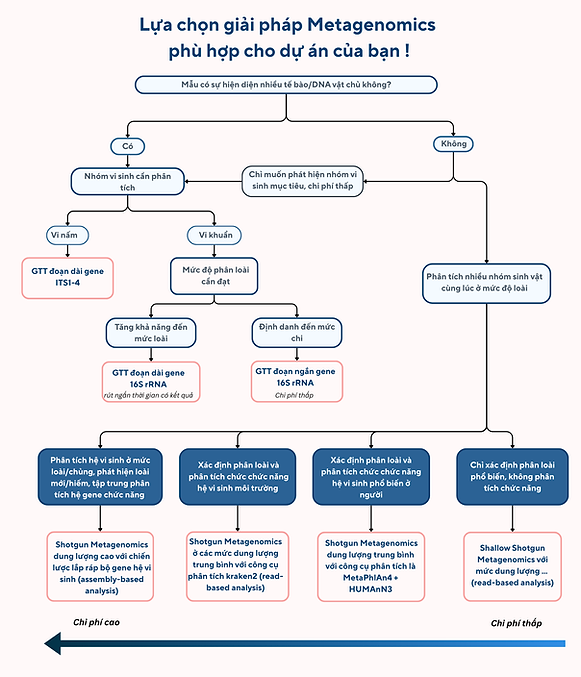
Metabarcoding
Giới thiệu:
Metabarcoding là giúp xác định thành phần quần xã vi sinh vật dựa trên giải trình tự vùng gene chỉ thị như 16S rRNA (vi khuẩn), ITS (nấm),.... Phương pháp này tiết kiệm chi phí và giúp nhanh chóng để hiểu cấu trúc của một cộng đồng vi sinh, tuy nhiên khả năng phân loại đến mức loài và hiểu về chức năng còn giới hạn và chỉ nhận diện nhóm vi sinh vật mục tiêu.
Nền tảng giải trình tự:
Giải trình tự đoạn ngắn 2 x 150bp hoặc giải đoạn dài MinION (Oxford Nanopore)
Phổ vi sinh vật phát hiện:
Vi khuẩn, vi khuẩn cổ với 16S rRNA và nấm với ITS
Kết quả nhận được:
- Thành phần và tỷ lệ hiện diện của hệ vi sinh mục tiêu
- Độ đa dạng của hệ vi sinh mục tiêu và giữa các mẫu
- Xác định nhóm sinh vật đặc trưng trong mẫu
Thời gian trả kết quả:
2 -3 tuần hoặc 4-6 tuần
Shotgun metagenomics
Giới thiệu:
Thay vì chỉ giải trình tự gene chỉ thị, shotgun metagenomics giải trình tự toàn bộ vật liệu truyền trong mẫu môi trường. Điều này không chỉ cho phép xác định thành phần, tỷ lệ vi sinh vật hiện diện trong mẫu mà còn cung cấp thông tin chú giải thành phần các gene chức năng. Phương pháp này cần lượng dữ liệu và chi phí cao hơn metabarcoding.
Nền tảng giải trình tự:
Giải trình tự đoạn ngắn 2 x 150 bp với đa dạng các mức dung lượng giải khác nhau.
Phổ vi sinh vật phát hiện:
Vi khuẩn, vi khuẩn cổ, DNA virus, nấm, eukaryote
Kết quả nhận được:
- Thành phần, tỷ lệ hiện diện và độ đa dạng của các loài
- Thành phần và tỷ lệ gene kháng kháng sinh
- Thành phần và tỷ lệ các con đường biến dưỡng (KEGG)
Thời gian trả kết quả:
4-6 tuần

QUÝ KHÁCH SẼ NHẬN ĐƯỢC
Tùy thuộc vào yêu cầu cụ thể, quý khách sẽ nhận được những kết quả phân tích cũng như dịch vụ khác nhau. Dưới đây là một số kết quả hoặc dịch vụ quý khách có thể nhận được:
-
Kết quả phân tích thành phần các loài có trong mẫu với các chỉ tiêu: sự hiện diện và phân loại các loài, tỷ lệ các loài, độ da đạng,…
-
Kết quả so sánh thành phần loài ở các mẫu hay nhóm mẫu (nếu có nhiều mẫu).
-
Phân tích các gene chức năng (con đường biến dưỡng, gene kháng kháng sinh,…) nếu phân tích shotgun metagenomics.
-
Báo cáo tóm tắt phương pháp, quy trình thực hiện.
-
Các kết quả phân tích khác theo yêu cầu khách hàng.
-
Tư vấn quy trình chuẩn bị mẫu, kết quả và tham gia công bố kết quả nếu khách hàng có yêu cầu.
QUY TRÌNH CHUNG

1. Nhận yêu cầu và tư vấn

2. Thu, nhận mẫu

3. Tách chiết DNA/RNA

4. Chuẩn bị thư viện

5. Giải trình tự

6. Phân tích dữ liệu

7. Trả kết quả và tư vấn





